微生物組與代謝組的數據整合方法按照是否基于已有知識可以分為統計學檢驗方法和知識驅動的整合分析方法。統計學檢驗方法采用單變量或多變量分析闡述不同組學層級所屬生物學指標之間的相關性。單變量相關分析分析過程相對簡單,假陽性率高,需要多重校正檢驗以控制Ⅰ型錯誤率,因此并不適用于分析多組學數據。多變量相關分析可以考慮到復雜樣本中多個指標的相關性,由于測序和質譜測量之間的測量單位不同,這種方法不太適用于微生物與代謝物的相互作用。知識驅動的整合分析方法是將單一組學層級獲取的生物學指標投射到已有數據庫中,以解釋各生物學指標之間的相互聯系,該方法包括常用的構建相關性網絡、種子集框架和通過預測代謝更新率構建代謝模型,從而將微生物群落與代謝物相關聯。
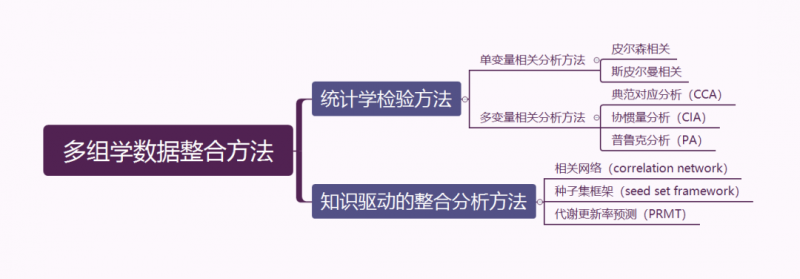
本片文章中將主要介紹伯豪現有的兩種微生物組與代謝組聯合分析方法——mmvec 和 MIMOSA 2。mmvec 主要考慮到微生物與代謝物的共現概率來預測可能的微生物與代謝物的相互作用;MIMOSA 2 對 PRMT 的功能進行了擴展,整合代謝組學數據研究微生物組成和代謝活性。這兩種分析方法對目前常用的聯合分析方法進行了進一步的補充,結果相對更加可信。
微生物與代謝組聯合分析方法 —— mmvec 神經元網絡介紹
2019 年 11 月 Rob Knight 團隊在《Nature Methods》發表了一篇“Learning representations of microbe-metabolite interactions”提出 mmvec (microbe-metabolite vectors) 神經元網絡,該方法通過學習代謝物和微生物共現概率(共現概率是指在觀察到微生物的情況下觀察代謝物的條件概率,從而使我們能夠確定可能的微生物與代謝物的相互作用),對代謝物與微生物互作進行評級,并可視化顯示分析結果。
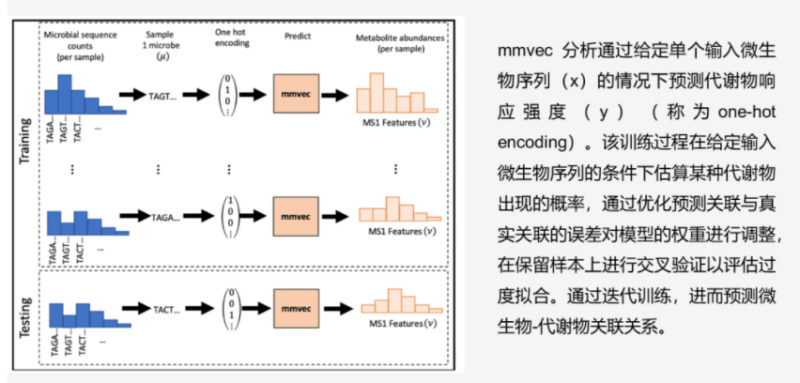
mmvec 分析原理示意圖

分析比較
文章中作者將 mmvec 與 Pearson,Spearman,SPIEC-EASI,SparCC 和 Proportionality 等分析方法進行了比較,證明 mmvec 的 F1 score、高精度和召回率更高,且該模型具有一定的魯棒性。同時,研究人員以已知的環境(沙漠土壤濕潤生物結殼)和臨床(囊性纖維化肺)實例為例,展示了這一方法恢復微生物與代謝物之間關系的能力,并證明了該方法如何發現微生物產生的代謝產物與炎癥性腸病之間的關系。
微生物與代謝組聯合分析方法 —— MIMOSA 2 介紹
2016 年 Borenstein Lab 提出 MIMOSA 模型,該方法對 PRMT 進行了擴展,通過預測微生物群落代謝能力——CMP(community-wide metabolite potential)構建一個代謝模型來預測群落組成對代謝物濃度的影響,并評估該預測值是否與測量到的代謝組學特征相符,從而將物種組成和代謝物濃度相聯系,該方法在 2018 年進行了升級——MIMOSA 2。
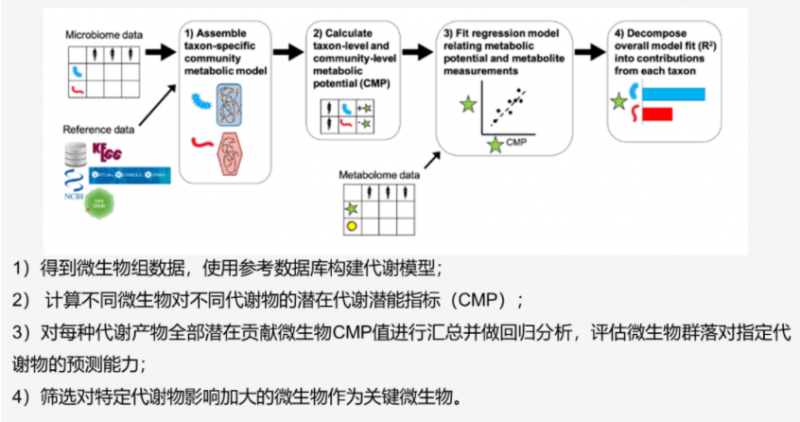
通過 MIMOSA 2 分析主要可以回答一下三個問題:
1)代謝組數據是否和微生物組數據緊密相關;
2)微生物群落差異是否可以解釋代謝差異;
3)何種微生物或者基因造成了代謝差異,或者貢獻了代謝差異。
結果展示
1. mmvec 神經元網絡主要結果展示
▲微生物和代謝物共現概率 biplot

▲微生物和代謝物共現概率 heatmap
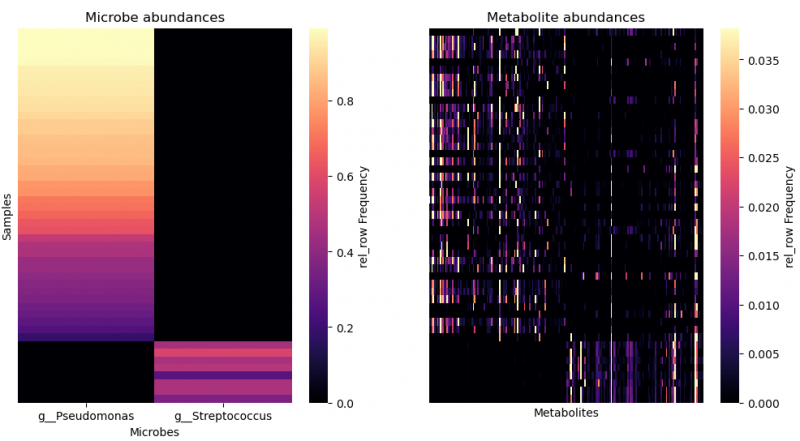
▲指定微生物及其共現代謝物 paired heatmap
2. MIMOSA 2 主要結果展示
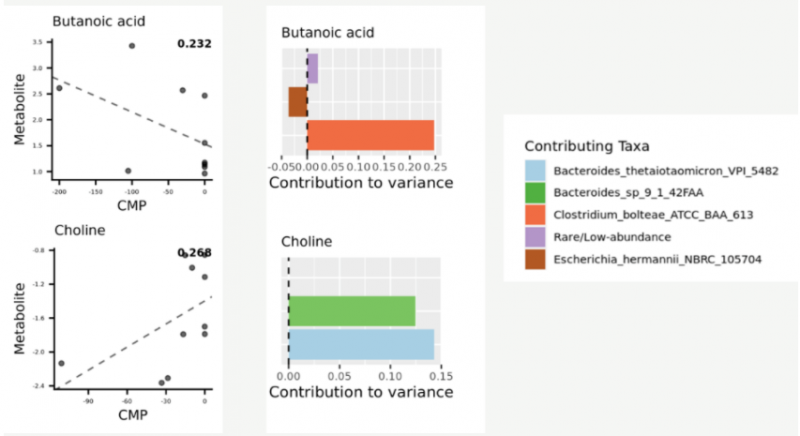
▲微生物對代謝物貢獻率結果展示
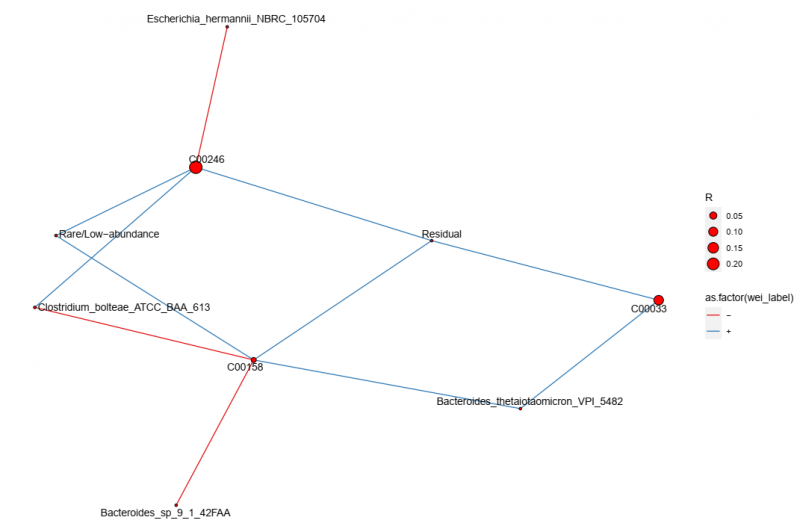
▲微生物與代謝物網絡圖展示
參考文獻:
1. 侯璐文,吳長新,秦雪梅,等。腸道微生物功能宏基因組學與代謝組學關聯分析方法研究進展 [J]. 微生物學報,2019, 059(009):1813-1822.
2. Morton J T , Aksenov A A , Nothias L F , et al. Learning representations of microbe–metabolite interactions[J]. Nature Methods, 2019, 16(12).
3. Noecker C , Eng A , Srinivasan S , et al. Metabolic Model-Based Integration of Microbiome Taxonomic and Metabolomic Profiles Elucidates Mechanistic Links between Ecological and Metabolic Variation[J]. Msystems, 2016, 1(1).
更多伯豪生物人工服務:


